「コロナウイルス2019 B.1.1.7 変異株に感染したマレートラ ―― アメリカ合衆国ヴァージニア州の事例」 『新興感染症』 2021年12月 27巻12号
2021 Oct 14; 27 (12). doi: 10.3201/eid2712.211234, Patrick K Mitchell, Mathias Martins, Tara Reilly et al., SARS-CoV-2 B.1.1.7 Variant Infection in Malayan Tigers, Virginia, USA - Volume 27, Number 12 — December 2021
Patrick K. Mitchell, Mathias Martins, Tara Reilly, Leonardo C. Caserta, Renee R. Anderson, Brittany D. Cronk, Julia Murphy, Erin L. Goodrich, and Diego G. Diel
『新興感染症』(Emerging Infectious Diseases)はアメリカ疾病予防管理センター(Centers for Disease Control and Prevention, CDC)が発行する雑誌で、本論文は同誌の2021年12月 27巻12号に掲載されたものである。本論文はヴァージニア州の動物園でマレートラに発生したコロナウイルス2019アルファ株(SARS-CoV-2 B.1.1.7)の集団感染を報告している。
広川による和訳を付して、以下に本文を示す。なお筆者(広川)の訳はこなれた日本語で分かりやすく記述することを重視したため、必ずしも逐語訳にはなっていない。文意を通じやすくするために補った語は、ブラケット [ ] で囲った。
| SARS-CoV-2 B.1.1.7 Variant Infection in Malayan Tigers, Virginia, USA | |||
| (コロナウイルス2019 B.1.1.7 変異株に感染したマレートラ ―― アメリカ合衆国ヴァージニア州の事例) | |||
| On April 4, 2021, a 5-year-old male Malayan tiger (Panthera tigris
jacksoni) at the Virginia Zoo (Norfolk, VA, USA) began exhibiting lethargy,
labored breathing, coughing, intermittent upper respiratory sounds, hyporexia,
and mucoid nasal discharge. On April 7, another 5-year-old male Malayan
tiger began experiencing labored breathing, cough, clear nasal discharge,
and hyporexia. On April 10, a third Malayan tiger, a 10-year-old male,
had cough and later clear nasal discharge. The tigers' clinical signs resolved
by April 15, eleven days after the outbreak began. |
2021年4月4日、ヴァージニア動物園(ヴァージニア州ノーフォーク)で飼育されている5歳のマレートラ雄(Panthera tigris jacksoni)に倦怠感、呼吸困難、咳、上部呼吸器系の間歇的雑音、食欲不振、粘液性の鼻水の症状が現われた。4月7日には別の5歳のマレートラ雄に呼吸困難、咳、透明な鼻水、食欲不振の症状が現れた。4月10日には10歳のマレートラ雄に咳が始め、やがて透明な鼻水の症状も現れた。これら三頭の臨床症状は集団発生から11日目にあたる4月15日までに収まった。 | ||
| Zoo staff collected nasal swab and fecal samples from the 5-year-old
tigers on April 9 and the 10-year-old tiger on April 13 and submitted these
to Cornell University's Animal Health Diagnostic Center (AHDC; Ithaca,
NY, USA). AHDC tested samples for Bordetella sp., Chlamydia felis, Mycoplasma cynos, M. felis, Streptococcus equi subspecies zooepidemicus, influenza virus, pneumovirus, feline calicivirus, and feline
herpesvirus; all results were negative. |
動物園では4月9日に5歳の二頭、4月13日に10歳の一頭からそれぞれ鼻腔拭い液と糞便検体を採取し、ニューヨーク州イサカにあるコーネル大学動物健康診断センター(AHDC)に提出した。AHDCではこれらの検体からボルデテラ属の各種細菌、猫クラミジア、犬マイコプラズマ、猫マイコプラズマ、ウマ連鎖球菌の一変種(S.
equi ssp. zooepidemicus)、インフルエンザウイルス、ニューモウイルス、猫カリシウイルス、猫ヘルペスウイルスの検出を試みたが、結果は全て陰性であった。 |
||
| All samples tested positive for severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) by EZ-SARS-CoV-2 Real-Time RT-PCR Test (Tetracore, Inc., https://tetracore.com). We isolated SARS-CoV-2 from respiratory and fecal specimens from the first tiger. Testing at the US Department of Agriculture National Veterinary Services Laboratories (Ames, IA, USA) confirmed SARS-CoV-2 infection. We screened the tiger samples using TaqPath COVID-19 RT-PCR Kit (Thermo Fisher Scientific, https://www.thermofisher.com), which revealed a spike gene dropout in samples from all 3 tigers; only the nucleoprotein and open reading frame 1ab gene targets were detected, suggesting B.1.1.7 variant infection. | EZコロナウイルス2019迅速逆転写PCR検査(テトラコア社 https://tetracore.com)を用いた検査では、全ての検体が陽性であった。最初のトラの呼吸器から採取した検体及び糞便の検体から、我々はコロナウイルス2019を分離した。コロナウイルス2019への感染は、農務省の国立獣医学研究所(NVSL アイオワ州エイムズ)によって確定された。トラから採取した検体はタックパスコロナウイルス2019迅速逆転写PCRキット(サーモ・フィッシャー・サイエンティフィック社 https://www.thermofisher.com)でスクリーニングを行ったところ、三頭すべての検体からスパイク蛋白質の遺伝子ひとつが欠失している一方で、核蛋白質と ORF1a 及び ORF1b の遺伝子のみが検出され、アルファ株(B.1.1.7)への感染が示唆された。 | ||
| We performed whole-genome sequencing on all samples by using MinION (Oxford Nanopore Technologies, https://nanoporetech.com), as previously described (1). We assembled reads using the ARTIC ncov-2019 protocol (ARTIC Network, https://artic.network) and Medaka (Oxford Nanopore Technologies) for variant calling. We obtained near-complete (29,702–29,710-bp) assemblies from all nasal swab specimens (GenBank accession nos. MZ305031–3) but no assemblies from fecal samples. We identified respiratory specimen genomes as lineage B.1.1.7 (Alpha variant) by using Pangolin version 2.4.2 (https://github.com/cov-lineages/pangolin). We used Nextstrain (https://nextstrain.org) for phylogenetic analysis of tiger-derived sequences and other B.1.1.7 sequences downloaded from GISAID (https://www.gisaid.org) on April 15, 2021 (2, 3). | 先に書いた通り(1)、我々はオックスフォード・ナノポア・テクノロジーズ社(https://nanoporetech.com)のミニオン(MinION)を使用し、全ての検体で全ゲノム配列の読み取りを行い、アーティック・ネットワーク(https://artic.network)の新型コロナウイルスゲノム解読プロトコル及びオックスフォード・ナノポア・テクノロジーズ社のメダカを使用して読み取りデータを集積し、変異型の呼称を決定した。全ての鼻腔拭い液検体からほぼ完全なゲノムアセンブリ(29,702ないし 29710塩基対)を得た(GenBank受け入れ番号MZ305031~305033)が、糞便サンプルからゲノムアセンブリは得られなかった。呼吸器に由来する検体のゲノムは、パンゴリン ver. 2.4.2(https://github.com/cov-lineages/pangolin)を使用して、B.1.1.7系統(アルファ変異株)と同定された。トラ由来のゲノム配列の系統分析には、ネクストストレイン(https://nextstrain.org)、及び 2021年4月15日にジゼイド(https://www.gisaid.org)からダウンロードした他の B.1.1.7系統ウイルスのゲノム配列を使った(2, 3)。 | ||
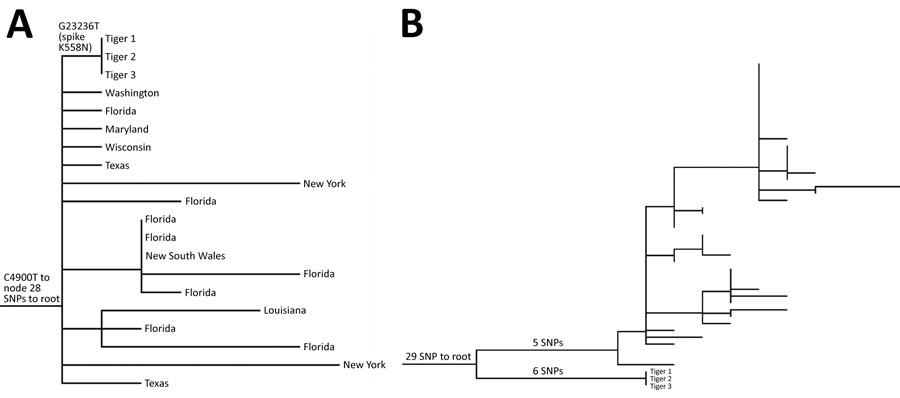
| Tiger-derived sequences all were identical, except 1 manually corrected homopolymer repeat error, and fell into a clade defined by a C4900T mutation containing other samples collected primarily in the United States. Tiger-derived sequences differed from others in the clade by 1 single-nucleotide polymorphism in the spike gene (K558N) (Figure, panel A). | トラ由来のゲノム配列は、単独重合体の複製エラー一か所を手動で修正したことを除けば全ての検体においてまったく同一であり、主にアメリカ合衆国内で集められた他の検体とともに、変異C4900Tで定義されるクレードと同定された。トラ由来のゲノム配列が同一クレードの他のものと異なっていたのは、スパイク遺伝子(K558N)における一か所の一塩基多型のみであった(図A)。 | ||
| Using the vdb tool (4), we found 46 additional B.1.1.7 sequences that had the K558N mutation in GISAID on July 22, 2021; all were collected from Virginia during March 27 – July 7, 2021. However, phylogenetic analysis of these sequences and the tiger-derived sequences showed divergence of 11 single-nucleotide polymorphism, minus the divergence producing the K558N mutation (Figure, panel B), indicating the sequences are not related epidemiologically. | 我々は変異型データベース(4)を使って B.1.1.7系統に属するウイルスのゲノム配列を精査し、2021年7月22日にジゼイドの中から K558Nの変異を有する46種の配列を見出した。それらは全て 2021年3月27日から7月7日の間にヴァージニア州で採集されたものであった。これらのゲノム配列とトラ由来のゲノム配列の間に成り立つ系統的発生関係を分析すると、11か所の一塩基多型に分岐することが分かった。しかしながらこれら11か所の一塩基多型は K558Nに繋がる分岐とはなっておらず(図B)、46種のゲノム配列とトラ由来のゲノム配列の間に疫学的関係は無いことが示された。 | ||
 |
|||
| Figure. Maximum-likelihood phylogenetic trees of severe acute respiratory syndrome coronavirus 2 from 3 Malayan tigers, Virginia, USA. Tiger samples are numbered in order of symptom onset. | 図 アメリカ合衆国ヴァージニア州のマレートラ三頭から分離されたコロナウイルス 2019に関して、最も蓋然性の高い系統樹。トラの検体に付けられた番号は、発症した順を示す。 | ||
| A) Subset of phylogenetic tree showing parent (G23236T) and grandparent
(C4900T) nodes of the tiger sequences, with tips labeled as states of origin
in the United States or Australia. B) Phylogenetic tree showing that other B.1.1.7 viruses detected in Virginia that contain the K558N mutation are not epidemiologically related to the sequences detected in tigers 1, 2, and 3. SNP, single-nucleotide polymorphism. |
・図Aはコロナウイルス2019アルファ株の系統樹の一部で、トラのウイルスに繋がるペアレントのノード(G23236T)及びグランドペアレントのノード(C4900T)を示す。各末端に表示されたアメリカ合衆国またはオーストラリアの州名は、各変異型が最初に検出された場所を示す。 ・図Bの系統樹は、ヴァージニアで検出され、K558Nの変異を含むアルファ株の各変異型が、三頭のトラから検出された型と疫学的に繋がらないことを示す。 ※ SNP は一塩基多型(single nucleotide polymorphisms)の略記。 |
||
| The source of the tigers' infection is unknown. The zoo has been open to the public, but transmission from a visitor is unlikely because tiger exhibit areas are separated from visitors by either a glass enclosure or >9 m distance. The most plausible explanation is that >1 tiger acquired the virus from a keeper because they had close contact. However, no employees tested positive for SARS-CoV-2 nor had symptoms during the 4 weeks before the tigers' symptom onset. Nine keepers were responsible for the animals' daily care; 2 other persons prepared animal diets daily. Employees were required to wear facemasks always, indoors and outdoors; everyone wore standard 2-ply surgical masks or homemade cloth facemasks. Staff also were required to wear gloves when handling and preparing food and when servicing animal areas. Furthermore, staff were required to step into an accelerated hydrogen peroxide disinfectant footbath when entering the tiger building and diet kitchen. The 3 tigers might have been infected by an employee, or 1 tiger was infected, then transmission occurred to the others. Two tigers lived in the same enclosure and had no direct contact with the third, but all 3 rotated through common enclosure spaces. | トラの感染源は不明である。動物園には大勢の人々が訪れていたが、トラの展示区域はガラスの囲いまたは九メートルを超える距離で観客から隔てられているゆえに、観客からウイルスを貰った可能性は考えにくかった。最もありそうな説明は二頭以上のトラが飼育係からウイルスを貰ったということである。なぜならトラは飼育係と濃厚接触していたからである。しかしながらコロナウイルス2019の検査で陽性になった職員も、トラが発症する四週間前から症状があった職員もいなかった。三頭のトラは、日常的な世話を九名の職員が、日々の食餌を他の二名の職員が、それぞれ担当していた。職員は就業規則によって屋内でも屋外でも常にマスクを着用していた。マスクは二枚重ねの標準的外科用マスク、または自作の布製マスクであった。食材の取り扱いと調理に際して、また動物の居住区画で作業をする際には、手袋を着用尾する規則であった。さらに職員がトラの建物と調理場に入る際は加速化過酸化水素の水槽に足を浸けて消毒する規則であった。トラたちは三頭とも一人の職員から感染した可能性もあるし、一頭が感染した後に他のトラに広まった可能性もある。トラのうちニ頭は同じ囲いに住んでおり、もう一頭のトラと直接的な接触は無かったが、すべてのトラは共通の囲い内を行き来していた。 | ||
| After identification of the tiger infections, 4 additional zoo animals
were tested: 1 lion (Panthera leo) with lethargy and hyporexia ≈1 week
after SARS-CoV-2 diagnosis in the tigers; another asymptomatic lion because
of age and proximity to the first lion; and 2 degus (Octodon degus) that
died in late March and had interstitial pneumonia on necropsy. AHDC tested
nasal swab samples from the lions and frozen spleen and cecum samples from
the degus by reverse transcription PCR; all results were negative for SARS-CoV-2. |
トラがコロナウイルス019に感染していることが判明した後、動物園では四頭の動物を追加で検査した。一頭のライオン(Panthera leo)は、トラがコロナウイルス2019感染症と診断されてから約一週間後に、倦怠感と食欲不振の症状を示した。もう一頭のライオンは無症状であったが、高齢であり、最初のライオンの近くで飼育されていたために、検査の対象となった。二頭のデグー(Octodon degus)は三月下旬に亡くなり、剖検により間質性肺炎が見つかった。ライオンの鼻腔拭い検体、及び冷凍保存されていたデグーの脾臓と盲腸はコーネル大学動物健康診断センター(AHDC)で逆転写PCR検査を受けた。検査結果はコロナウイルス2019に関してすべて陰性であった。 | ||
| Our findings underscore felid susceptibility to SARS-CoV-2, which also has been detected in captive snow leopards (Panthera uncia) and pumas (Puma concolor) (5). Other nonhuman species, including gorillas (Gorilla gorilla), minks (Neovison vison), and ferrets (Mustela putorius furo), have acquired SARS-CoV-2; additional species have been shown to be susceptible experimentally (5 –7). Domestic cats and dogs in the United Kingdom and United States reportedly had B.1.1.7 infections, suggesting that mutations characterizing this lineage are not constrained to a host range (8; L. Ferasin et al., unpub. data, https://doi.org/10.1101/2021.03.18.435945). Monitoring animals for SARS-CoV-2 infection is critical to determining potential host range, particularly as new virus variants emerge and spread. | ネコ科動物はコロナウイルス2019に感受性があると考えられているが、我々が得た結果もその知見を裏書きしている。コロナウイルス2019は飼育下のユキヒョウ(Panthera uncia)とピューマ(Puma concolor)からも検出されている(5)。ゴリラ(Gorilla gorilla)、ミンク(Neovison vison)、イタチ(Mustela putorius furo)をはじめ、人間以外の種もこれまでコロナウイルス2019に感染しており、さらに別の数種に関しても実験によって感受性が示されている(5 –7)。イギリスとアメリカ合衆国の家庭で飼われている犬猫には、コロナウイルス 2019アルファ株(B.1.1.7)への感染が見出されたと報告されているが、このことはアルファ株を特徴づける突然変異が宿主域のみに限定されないことを示している(8; L. Ferasin et al., unpub. data, https://doi.org/10.1101/2021.03.18.435945)。動物のコロナウイルス2019感染を監視することは、とりわけ新しいウイルス変異株が現われて拡散する際に、宿主域となりうる動物種の範囲を確定するうえで極めて重要である。 | ||
| Dr. Mitchell is a research associate in the Department of Population Medicine and Diagnostic Sciences at Cornell University. His primary research interest is molecular epidemiology of infectious diseases. | ミッチェル博士はコーネル大学健康増進医学部助手であり、感染症の分子疫学を主に研究している。 | ||
| Acknowledgments | 謝辞 | ||
| This article was preprinted at https://doi.org/10.21203/rs.3.rs-618448/v1. |
本記事はピア・レヴューに先立ち https://doi.org/10.21203/rs.3.rs-618448/v1 で公開された。 | ||
References
| 1. | C Caserta L, Mitchell PK, Plocharczyk E, Diel DG. Identification of a SARS-CoV-2 lineage B1.1.7 virus in New York following return travel from the United Kingdom Microbiol Resour Announc. 2021; 10: e00097–21. | ||
| 2. | Hadfield J, Megill C, Bell SM, Huddleston J, Potter B, Callender C, et al. Nextstrain: real-time tracking of pathogen evolution. Bioinformatics. 2018; 34: 4121–3. | ||
| 3. | Sagulenko P, Puller V, Neher RA. TreeTime: Maximum-likelihood phylodynamic analysis. Virus Evol. 2018; 4. vex042. | ||
| 4. | West AP Jr, Wertheim JO, Wang JC, Vasylyeva TI, Havens JL, Chowdhury MA, et al. Detection and characterization of the SARS-CoV-2 lineage B.1.526 in New York. Nat Commun. 2021; 12: 4886. | ||
| 5. | World Organisation for Animal Health. COVID-19 [cited 2021 May 19]. https://www.oie.int/en/what-we-offer/emergency-and-resilience/covid-19 | ||
| 6. | US Department of Agriculture Animal and Plant Health Inspection Service. Cases of SARS-CoV-2 in animals in the United States [cited 2021 May 19]. https://www.aphis.usda.gov/aphis/dashboards/tableau/sars-dashboard | ||
| 7. | Delahay RJ, de la Fuente J, Smith GC, Sharun K, Snary EL, Flores Girón L, et al. Assessing the risks of SARS-CoV-2 in wildlife. One Health Outlook. 2021; 3: 7 | ||
| 8. | Hamer SA, Ghai RR, Zecca IB, Auckland LD, Roundy CM, Davila E, et al. SARS‐CoV‐2 B.1.1.7 variant of concern detected in a pet dog and cat after exposure to a person with COVID‐19, USA. Transbound Emerg Dis. 2021 May 12 [Epub ahead of print] |
Figure
Article Citations
Highlight and copy the desired format.
| EID | Mitchell PK, Martins M, Reilly T, Caserta LC, Anderson RR, Cronk BD, et al. SARS-CoV-2 B.1.1.7 Variant Infection in Malayan Tigers, Virginia, USA. Emerg Infect Dis. 2021;27(12):3171-3173. https://doi.org/10.3201/eid2712.211234 |
|---|---|
| AMA | Mitchell PK, Martins M, Reilly T, et al. SARS-CoV-2 B.1.1.7 Variant Infection in Malayan Tigers, Virginia, USA. Emerging Infectious Diseases. 2021;27(12):3171-3173. doi:10.3201/eid2712.211234. |
| APA | Mitchell, P. K., Martins, M., Reilly, T., Caserta, L. C., Anderson, R. R., Cronk, B. D....Diel, D. G. (2021). SARS-CoV-2 B.1.1.7 Variant Infection in Malayan Tigers, Virginia, USA. Emerging Infectious Diseases, 27(12), 3171-3173. https://doi.org/10.3201/eid2712.211234. |
DOI: 10.3201/eid2712.211234
Original Publication Date: October 14, 2021
Table of Contents – Volume 27, Number 12—December 2021
アカデミア神戸 医療関係資料 インデックスに戻る
アカデミア神戸 大人のための教養講座 インデックスに移動する
アカデミア神戸 トップページに移動する
Ἀναστασία ἡ Οὐτοπία τῶν αἰλούρων ANASTASIA KOBENSIS, ANTIQUARUM RERUM LOCUS NON INVENIENDUS